Introducción
La resistencia bacteriana se ha convertido en una situación problemática y emergente de alta complejidad, siendo una amenaza cada vez mayor para la salud pública y repercutiendo en el control de infecciones intrahospitalarias 1-3. El uso indebido y excesivo de los antibióticos es una de las principales razones por las cuales ha aumentado el número de reportes de resistencia, cobrando importancia a nivel médico y económico debido a las altas tasas de morbilidad y mortalidad. Esto se debe a las diversas y cada vez más eficaces formas que tienen los microorganismos para evadir la acción de los antibióticos, prolongando la estancia hospitalaria y aumentando los costos asistenciales 1,4,5.
Las bacterias han adquirido resistencia mediante la transferencia horizontal de genes, que ocurre sobre plásmidos, transposones y mutaciones. Las carbapenemasas son enzimas versátiles con un amplio espectro que hidrolizan la mayoría de los betalactámicos y son resistentes a la acción de los inhibidores. Las especies productoras de carbapenemasas más frecuentemente aisladas son Klebsiella pneumoniae, Escherichia coli y Gram negativos no fermentadores como Acinetobacter baumannii y Pseudomonas aeruginosa2. La carbapenemasa más frecuente es la enzima KPC y ha sido reportada en varios países de los cinco continentes. La KPC se encuentra con mayor frecuencia en el clon epidémico Klebsiella pneumoniae ST258, entre otras Enterobacteriaceae6.
Después del primer aislamiento clínico de K. pneumoniae resistente a carbapenémicos mediante la producción de carbapenemasa tipo KPC en Carolina del Norte, Estados Unidos, en 1996, su diseminación aumentó alrededor del mundo, ocasionando una disminución de los tratamientos óptimos y un aumento en las tasas de mortalidad. Por esta razón, caracterizar la resistencia se ha convertido en una prioridad con el propósito de definir terapias adecuadas 7. En Colombia, el problema es evidente gracias a las investigaciones realizadas por grupos dedicados a la prevención, vigilancia y control de las infecciones asociadas a la atención en salud (IAAS) y resistencia a los antimicrobianos 8,9.
Los carbapenémicos, debido a su amplio espectro de actividad, son efectivos contra las bacterias Gram negativas y generalmente se consideran un recurso para tratar infecciones multirresistentes, ya que se asocian con una alta mortalidad, un alto potencial de diseminación y altos costos de hospitalización. Como consecuencia, limitan drásticamente las opciones terapéuticas, siendo el tratamiento empírico inadecuado en la mayoría de los casos 10.
Algunos países, como Argentina y Colombia, han experimentado endemicidad en enzimas tipo KPC, mientras que países como Nueva Zelanda y Canadá solo tienen casos importados 10,11. Desde el primer reporte en el año 2006 en Colombia, se ha detectado su diseminación a casi todas las enterobacterias en varias ciudades del país, denotando que las variantes que presentan un mayor número son la KPC-2 y KPC-3. Por lo cual, la resistencia debe ser un tema prioritario en el territorio 12-14.
Aunque la resistencia a los carbapenémicos es una problemática en el ámbito mundial, su comportamiento varía geográfica y temporalmente, lo que hace importante vigilar en el ámbito local y nacional con el fin de evaluar el impacto que ocasionan las medidas que se implementen para la prevención y el control de resistencia a los antibióticos y las IAAS. Estas medidas se han implementado en muchas regiones del país, pero en el Caribe colombiano, específicamente en Barranquilla y Valledupar, no se registran reportes que contribuyan a la vigilancia de esta problemática, lo que conlleva a no tener registros reales. Por lo tanto, es necesario fortalecer la capacidad de detección y control con el objetivo de disminuir los casos de morbimortalidad y los costos hospitalarios.
En este contexto, este estudio tiene como propósito determinar la frecuencia de K. pneumoniae y E. coli productoras de carbapenemasas aisladas en cuatro instituciones de salud de Barranquilla y Valledupar.
Metodología
Tipo de estudio
Se llevó a cabo un estudio descriptivo de corte transversal retrospectivo para la detección molecular de la frecuencia de genes de carbapenemasas en K. pneumoniae y E. coli entre marzo de 2019 y marzo de 2020, aisladas de pacientes hospitalizados en cuatro instituciones de salud de Barranquilla y Valledupar. Se recibieron todos los aislamientos de las instituciones participantes. No se realizó el cálculo del tamaño de las muestras debido a que se trató de un estudio poblacional.
Se incluyeron los aislamientos de K. pneumoniae y E. coli que presentaron resistencia o resistencia intermedia a uno o más carbapenémicos. Se excluyeron los aislamientos que reportaron sensibilidad a los carbapenémicos y que no fueron procesados en los laboratorios de las instituciones participantes.
Recolección de los aislamientos
Cada aislamiento entregado por las instituciones venía acompañado de una ficha técnica que contenía información microbiológica detallada y fue registrada en una base de datos tipo Excel. El antibiograma fue realizado mediante la técnica de concentración inhibitoria mínima (CIM) utilizando los equipos Phoenix o Vitek.
Los aislamientos almacenados fueron reconstituidos tomando 10 µl de la muestra con un asa en aro calibrada y sembrados por agotamiento en agar Mueller Hinton. Posteriormente, se incubaron durante 24 horas con el objetivo de permitir el crecimiento del microorganismo y verificar la pureza del aislamiento.
Para la caracterización genotípica, los aislamientos fueron enviados al Laboratorio de Biología Molecular de la Universidad Metropolitana. Con fines de conservación, los aislamientos de K. pneumoniae y E. coli se depositaron en viales de 2 ml que contenían 1 ml de caldo tioglicolato suplementado con glicerol al 20 %, y se almacenaron a -80 °C en un ultracongelador 15.
Extracción de ADN de los aislamientos
La extracción de ADN bacteriano se realizó siguiendo el siguiente protocolo estandarizado: se suspendieron más de 30 colonias bacterianas en 1 ml de buffer Tris-HCL en un vial de 1,5 ml, agitándolas en un vortex durante 30 segundos. A continuación, se centrifugó a 14.000 rpm durante 2 minutos, se retiró el sobrenadante y se añadieron 300 µL de la Solución de Lisis de núcleo, mezclando suavemente hasta que las células se resuspendieron. La muestra se incubó a 80 °C durante 5 minutos en una plancha de calentamiento seco.
Posteriormente, se añadieron 1,5 µL de Solución RNasa, se invirtió el tubo 5 veces y se incubó a 37 °C durante 30 minutos en un baño serológico. Se permitió que la muestra se enfriara a temperatura ambiente y se adicionaron 100 µL de solución precipitadora de proteínas. Se agitó a alta velocidad en un vortex durante 20 segundos y luego se incubó la muestra en hielo durante 5 minutos. Posteriormente, se centrifugó a 14.000 rpm durante 3 minutos, transfiriendo el sobrenadante que contenía el ADN a un nuevo vial de 1,5 ml que contenía 300 µL de isopropanol a temperatura ambiente.
Se mezcló suavemente por inversión hasta que se visualizó una masa o filamentos de ADN. Después, se centrifugó a 14.000 rpm durante 2 minutos. Se eliminó cuidadosamente el sobrenadante y se dejó secar sobre papel absorbente con el tubo invertido. Se añadieron 300 µL de etanol al 70 %, mezclándolos suavemente por inversión varias veces para lavar el pellet de ADN. Posteriormente, se centrifugó a 14.000 rpm durante 2 minutos, se invirtió el tubo sobre papel absorbente durante 15 minutos y se añadieron 50 µL de Solución de Rehidratación de ADN. La muestra se almacenó en la nevera a temperaturas entre 2 y 8 °C hasta su posterior utilización.
Detección molecular de los tipos de carbapenemasas
La detección de los tres tipos de carbapenemasas clasificadas según Ambler se llevó a cabo siguiendo el protocolo del Laboratorio de Genética Molecular Bacteriana de una institución universitaria (3, 6). Se utilizó un vial de 1,5 ml para preparar la mezcla maestra, la cual contenía Solución Buffer, dNTPs, MgCl2 y DNA polimerasa. La cantidad de mezcla maestra fue de 12,5 µL, a la que se le añadieron 0,625 µL de cada juego de cebadores (Tabla 1) y agua bidestilada Milli Q hasta alcanzar un volumen final de 25 µL por reacción. Se agregaron 23 µL de la mezcla y 2 µL de DNA de la muestra a cada vial.
Posteriormente, los viales se colocaron en el termociclador modelo T100 (Bio-Rad) previamente programado con las siguientes condiciones: desnaturalización inicial a 94 °C durante 5 minutos, seguido de 34 ciclos repetitivos de desnaturalización a 94 °C por 45 segundos, anillamiento a 53 °C por 45 segundos, elongación a 72 °C por 1 minuto y, finalmente, una conservación a 8 °C. La visualización del producto de PCR se realizó mediante un gel de agarosa al 1 % con Syber Green, utilizando el fotodocumentador Gel-Doc (Bio-Rad).
Tabla 1
Cebadores utilizados para la detección de carbapenemasas
Resultados
Se recolectó un total de 86 aislamientos clínicos entre marzo de 2019 y marzo de 2020. De estos, el 69,77 % (60/86) provino de Barranquilla, mientras que el 30,23 % (26/86) se originó en Valledupar. En relación con los sitios de infección, la distribución porcentual según el tipo de muestra fue la siguiente: 36,66 % de orina, 33,33 % de sangre, 20 % de secreción bronquial y 10 % de líquido biliar (ver Figura 1).
Figura 1
Distribución porcentual de los aislamientos según el sitio de infección anatómico del paciente. Fuente: elaboración propia.

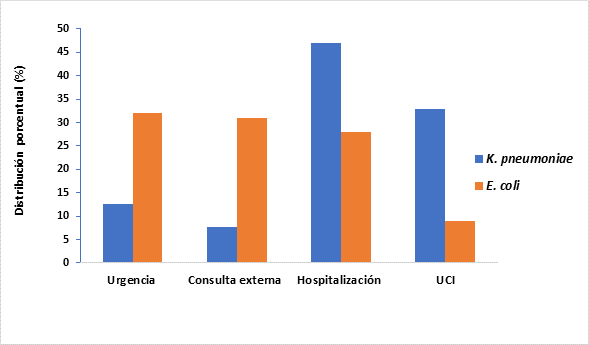
En cuanto a su distribución por servicio hospitalario, la E. coli se recuperó en un 32 % de urgencias, 31 % de consulta externa, 28 % de hospitalizados y 9 % de UCI; mientras que en la K. pneumoniae fue lo contrario: se recuperó el 46,87 % de hospitalizados, el 32,81 % de UCI, el 12,50 % de urgencias y el 7,81 % de consulta externa (ver Figura 2).
Figura 2
Distribución porcentual de los aislamientos por servicio hospitalario de E. coli y K. pneumoniae. Fuente: elaboración propia.
Genotipificación de carbapenemasas
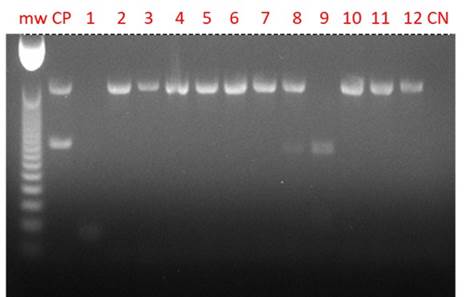
De los 86 aislamientos procesados mediante PCR, se pudo detectar el gen blaKPC en el 88,37 % (76/86) de los aislados. En el 1,16 % (1/86) se identificó el gen blaVIM, y el 10,47 % (9/86) restante fue confirmado como negativo para los genes de carbapenemasas probados. De los 76 aislados positivos para el gen blaKPC, el 81,58 % (62/76) correspondía a K. pneumoniae y el 18,42 % (14/76) fueron identificados como E. coli. Dentro de estos mismos aislados, el 76,31 % provenía de Barranquilla y el 23,69 % de Valledupar. La genotipificación se presenta en la Figura 3.
Figura 3
Genotipificación de Carbapenemasas. Carril mw: Marcador de peso molecular 50pb; CP: Control Positivo para gen blaKPC y para gen blaVIM: Carril 1: Aislamiento negativo. Carriles 2-8: Aislamiento positivo para gen blaKPC. Carril 9: Aislamiento positivo para gen blaVIM. Carriles 10-12: Aislamiento positivo para gen blaKPC. CN: Control negativo. Fuente: elaboración propia.
Discusión
Colombia se considera actualmente un país endémico en bacterias productoras de carbapenemasas. En esta investigación se evidenció que K. pneumoniae y E. coli son los aislados más frecuentes, con un 77,9 % (67/86) y 22 % (19/86) respectivamente. Asimismo, el tipo de muestra más frecuente de esos aislamientos fue orina (36,66 %) y sangre (33,33 %), resultados muy semejantes al estudio realizado por Guerra E. et al. (16, en el que la K. pneumoniae se aisló con mayor frecuencia en un 81 % (134/165) y 69 % (82/118) en dos años consecutivos. El tipo de muestra más frecuente fue sangre en un 41 % y orina con un 26 % 16.
Según lo reportado por Ovalle M. et al.17, se observó en Colombia un aumento en la resistencia a carbapenémicos en la K. pneumoniae en UCI, pasando del 9,3 % al 14 %. Además, en ese mismo estudio se encontró que el 70,3 % eran del tipo KPC, siendo la más identificada en enterobacterias, resultados similares a los reportados en este estudio. Otro estudio indica que la resistencia de microorganismos asociados a la atención en salud es de 14,4 % en la E. coli y 15 % en la K. pneumoniae en UCI y 13 % en no UCI; siendo estos resultados muy inferiores a los reportados en este estudio 18.
En cuanto a la genotipificación de las carbapenemasas, este estudio encontró que para la K. pneumoniae, un 88,37 % de los aislamientos presentaban el gen blaKPC, porcentaje menor que el reportado por Yauri M. et al.19, quienes al estudiar la diseminación clonal en 30 aislados bacterianos de este mismo microorganismo, encontraron un 100 % para la presencia del gen tipo blaKPC.
Según el grupo de vigilancia del Instituto Nacional de Salud (INS), al publicar los resultados de la vigilancia nacional de la resistencia antimicrobiana de enterobacterias y bacilos Gram negativos no fermentadores en infecciones asociadas a la atención de salud, Colombia se considera una región endémica para las enzimas KPC, con una frecuencia que alcanza el 70,3 % en especies de enterobacterias. De este grupo, la K. pneumoniae y la E. coli fueron los aislados más frecuentes en el servicio de UCI y hospitalización 17, resultados muy coherentes con esta investigación.
Por otro lado, en un estudio realizado en hospitales de la ciudad de Bogotá para determinar la diseminación de la K. pneumoniae, se reportó que de los 82 aislados, todos amplificaron para el gen tipo blaKPC. Asimismo, las muestras biológicas en las que se recuperó este microorganismo fueron orina (32,8 %) y sangre (25,6 %), resultados muy semejantes a este estudio 3.
El Laboratorio de Salud Pública del Atlántico realizó un estudio en instituciones de salud de Barranquilla, generando como resultado que las carbapenemasas tipo KPC fueron más frecuentes entre las enterobacterias (27,6 %), sola y asociada a otras carbapenemasas, predominando la K. pneumoniae (29,3 %) y la E. coli (9,6 %). Estos resultados difieren un poco de este estudio en porcentajes en relación con la K. pneumoniae, ya que en este estudio fue del 77,9 % y la E. coli de 22,1 %, pero desde el punto de vista de la prevalencia existe concordancia. Igualmente, hay semejanza entre los resultados de estos dos estudios en cuanto a la frecuencia de estos dos microorganismos en los servicios de hospitalización no UCI y el tipo de muestra biológica (orina), donde el más aislado fue la K. pneumoniae. Cabe destacar que para este mismo microorganismo en ambos estudios se detectó la presencia de carbapenemasa tipo blaVIM20.
En un estudio realizado por Valencia 20, en el que se llevó a cabo una descripción molecular de la E. coli productoras de carbapenemasas, se informó que el 75 % de los aislamientos presentaban el gen tipo blaKPC. La muestra de orina fue la más frecuente para el aislamiento del microorganismo productor de la enzima, con resultados superiores a los evidenciados en este trabajo, el cual registra un 18,42 % para el gen blaKPC en este mismo microorganismo, siendo la orina el tipo de muestra más frecuente. De manera similar, en ambos estudios se determinó que el mayor número de aislados bacterianos productores de carbapenemasas se obtuvo de los servicios clínicos de hospitalización, con el 44 % y el 46,87 %, respectivamente 21.
Existen diferencias epidemiológicas entre los países en cuanto a la frecuencia de los genes de resistencia en los microorganismos, como se evidencia en diversas publicaciones, como un estudio sobre la caracterización molecular de carbapenemasas en bacilos Gram negativos, en el que se reporta un 8 % para el gen blaKPC en enterobacterias, siendo K. pneumoniae el microorganismo predominante. Este resultado difiere significativamente del encontrado en este estudio, en el que el porcentaje total determinado del gen blaKPC es del 77,9 % 22.
En el mismo contexto, Requena et al.23 realizaron un estudio para la detección fenotípica y genotípica de carbapenemasas en enterobacterias aisladas en un laboratorio clínico. Encontraron que la muestra biológica más prevalente fue la orina, de la cual se aislaron los géneros bacterianos E. coli y K. pneumoniae, con el 61,3 % y el 14,9 %, respectivamente. Estos resultados son similares a los obtenidos en este estudio; sin embargo, en esa investigación se determinó que el 12,5 % (2/16) de los aislados eran portadores del gen tipo blaKPC, a diferencia de lo registrado en este estudio 23.
En este estudio, la resistencia a carbapenémicos en las instituciones fue del 89,53 % (blaKPC 88,37 % y blaVIM 1,16 %), y el 10,47 % restante fue confirmado como negativo para los carbapenémicos utilizados. Por lo tanto, se infiere que algunas razones para que las muestras dieran resultados negativos por PCR podrían ser: resistencia producida por un gen distinto a los detectados en el estudio o que la resistencia sea por mecanismos distintos a la producción de carbapenemasas, como bombas de eflujo, mutaciones de porinas, otras enzimas que hidrolizan beta lactámicos BLEE y AMPc. Por otro lado, la K. pneumoniae fue el microorganismo más reportado con un 77,9 %, principalmente en hospitalización (46,87 %) y UCI (32,81 %), lo que refleja que por parte de esta bacteria existe una afinidad por el ámbito hospitalario. Por lo anterior, se debe implementar la vigilancia de estos eventos, lo cual permitirá fortalecer la capacidad de detección y control de resistencia bacteriana dentro de las instituciones de salud, lo que permitirá caracterizar el perfil epidemiológico.